行业动态
通过突变解析致病RNA分析 用于作物病害的精确田间分子诊断
-
来源:
海南微氪生物科技股份有限公司
日期:2024-01-21 14:13:26
点击:332
属于:行业动态
通过突变解析致病RNA分析用于作物病害的精确田间分子诊断
1.文献摘要作物病害的分子诊断可以指导农药的精确施用,从而减少农药的使用,同时提高作物产量,但缺乏工具。研究人员提出了一种现场分子诊断工具,该工具使用廉价的比色纸和智能手机,可以多路、低成本、快速检测作物病原体。通过结合toehold介导的链置换和金属离子介导的脲酶催化反应,可实现病原RNA的快速无核酸扩增检测。研究人员对六种小麦病原真菌进行了多重检测,并对小麦条锈病进行了早期检测。当与用于快速核酸提取的微针和用于结果分析的智能手机应用程序相结合时,可以在大约10分钟内完成样本到结果的测试 。重要的是,通过检测真菌RNA和突变,该方法可以区分活的和死的病原体,并敏感地识别携带突变的抗真菌药菌株,为精确的作物病害管理提供基本信息。
2.文献内容
2.1分析原理
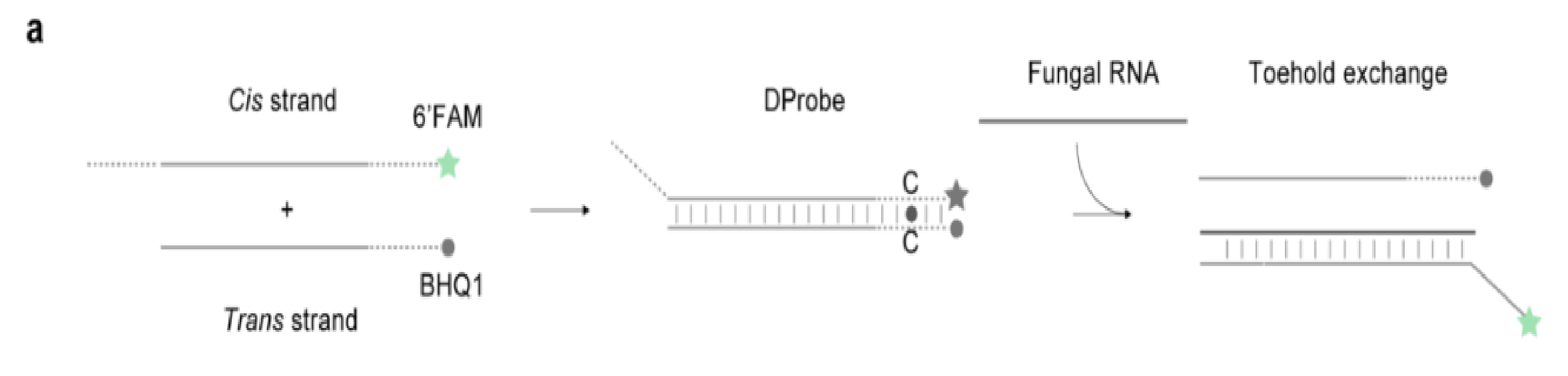
真菌RNA序列是根据简单的核酸反应识别的,称为toehold-mediated strand displacement(TMSD),需要双链DNA探针(DProbe) (图1)。
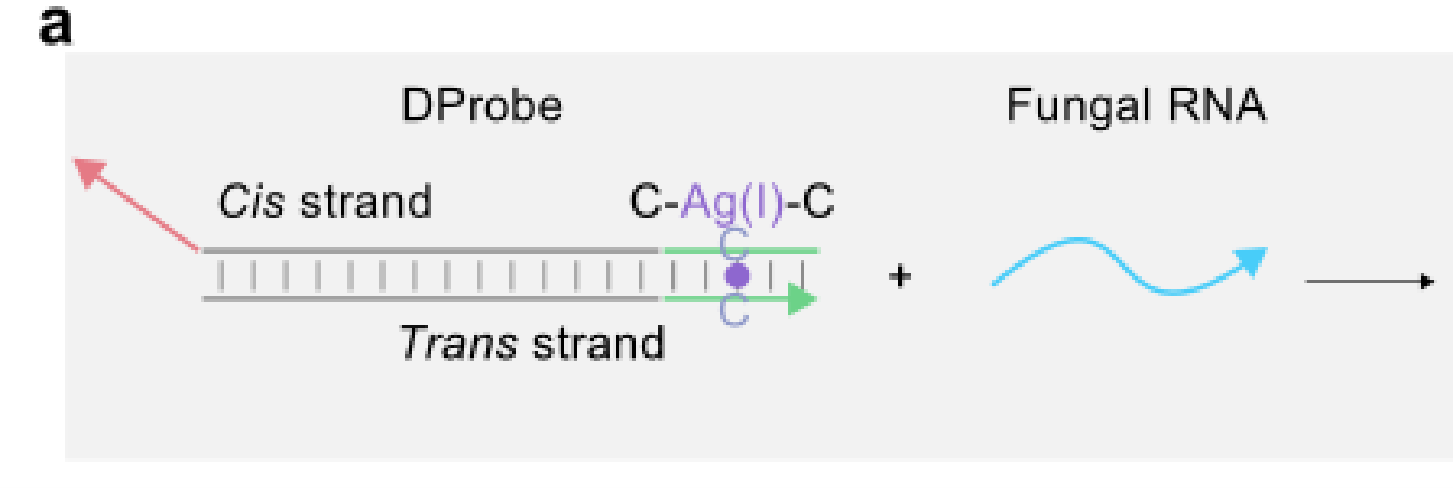
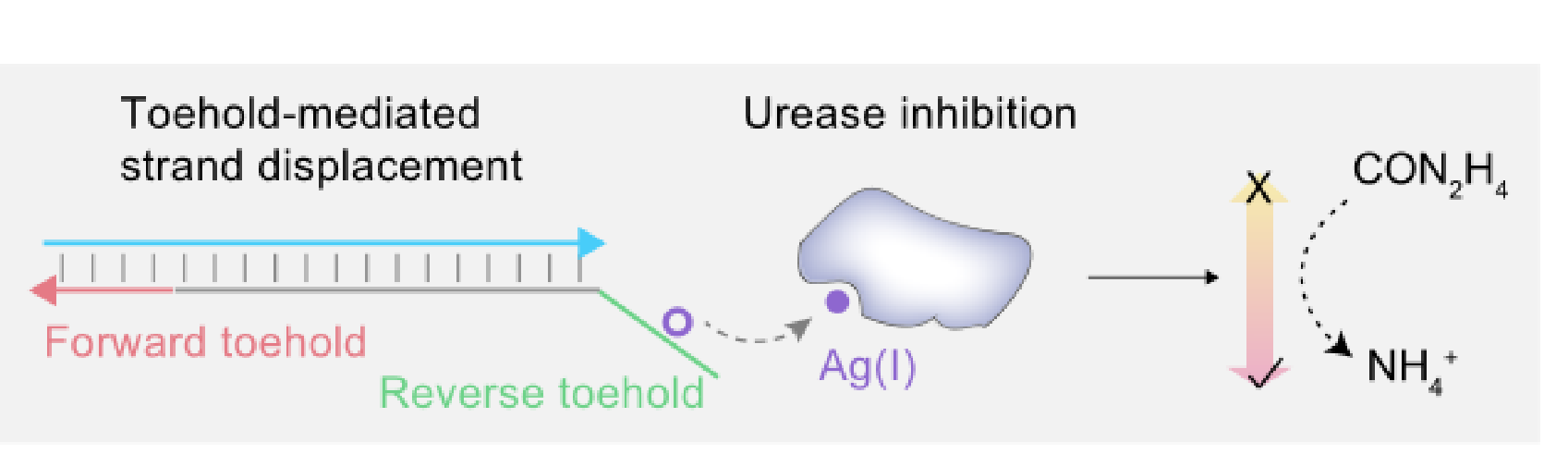
图1|比色分析示意图
DProbe是一种双链DNA,顺式链和反式链的混合体。真菌RNA为TMSD反应提供染料。TMSD反应的驱动力来自前趾(粉红色区域)悬突和真菌RNA之间的杂交,阻力来自反向趾(绿色区域)杂交的破坏。真菌RNA诱导释放的游离Ag(I)离子抑制脲酶活性,减少NH的生成 four+根据这一原理,可以通过pH指示剂酚红的颜色变化来检测真菌RNA的存在。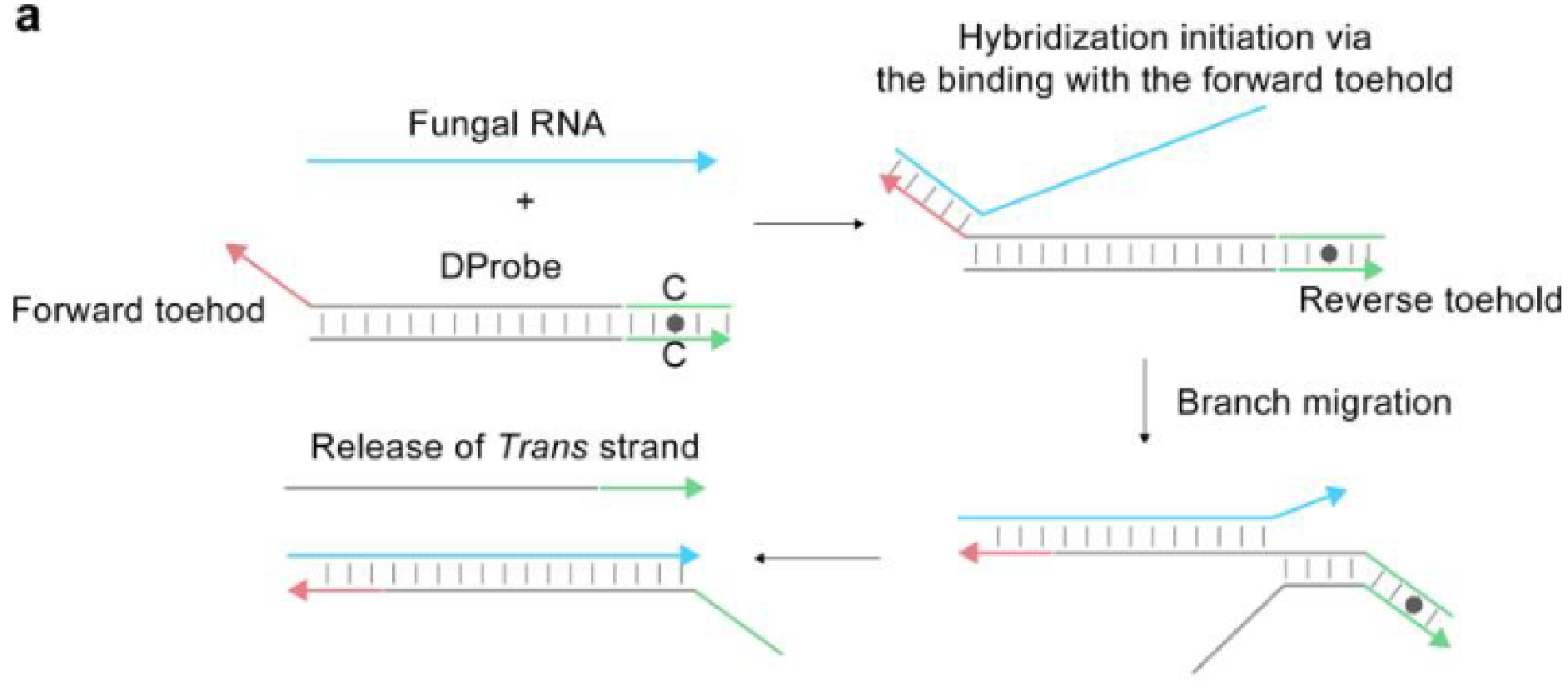
图2|TMSD反应
在DProbe中,顺式链与较短的序列(反式链)杂交,产生单链悬突结构域(粉红色)。真菌RNA与悬突结构域结合,形成“立足点”,迫使与顺式链杂交1,2。真菌RNA和Cis链的顺序杂交以及DProbe杂交体的断裂产生分支迁移。最终,反式链仅与顺式链中与真菌RNA不互补的短结构域(反向立足点)结合,并脱离来自Cis链。股线位移过程的净效应由前趾内新碱基对的形成和后趾内前碱基对的破坏决定。通过调节DProbe中的趾端结构域,TMSD允许基于它们对Cis链的不同亲和力产生对非靶RNA结合的阻碍和对靶核糖核酸结合的允许。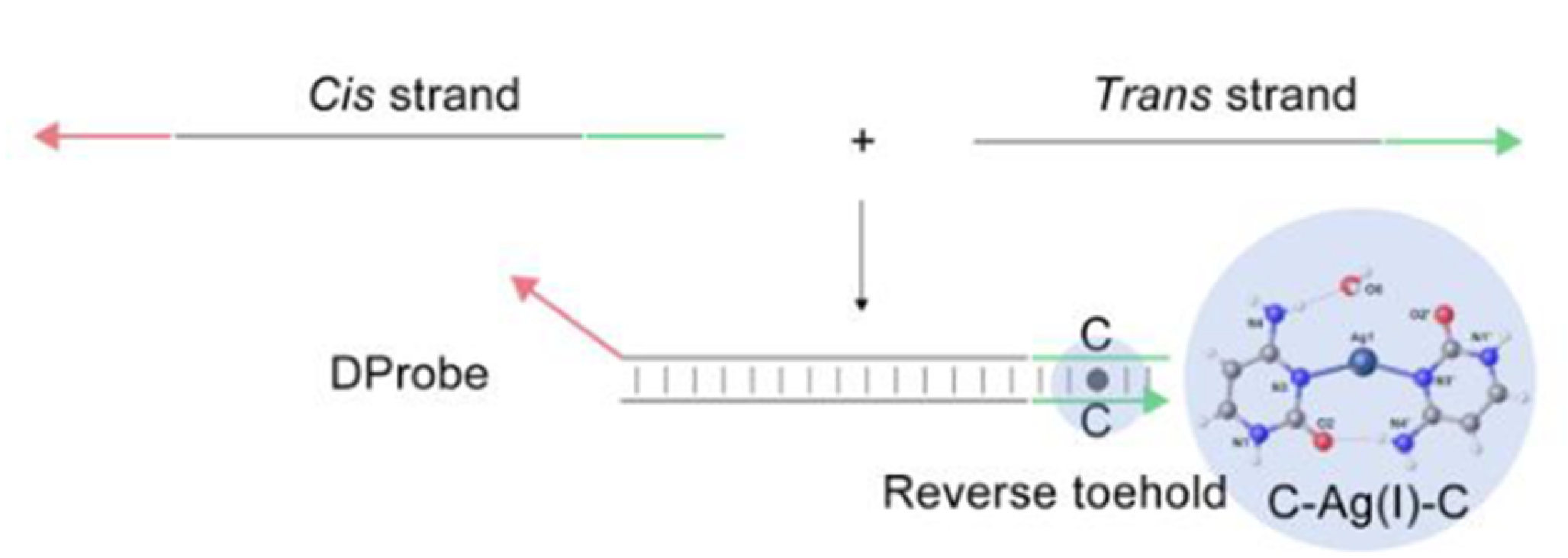
图3|胞嘧啶Ag(I)-胞嘧啶人工碱基对
Ag(I)离子结合在胞嘧啶-胞嘧啶错配对中的N3个胞嘧啶氮原子,形成胞嘧啶Ag(I)-胞嘧啶(C-Ag(I)-C)人工碱基对3。通过在反向脚趾处设计C-C失配区域,Ag(I)离子允许通过形成人工碱基对插入到DProbe中。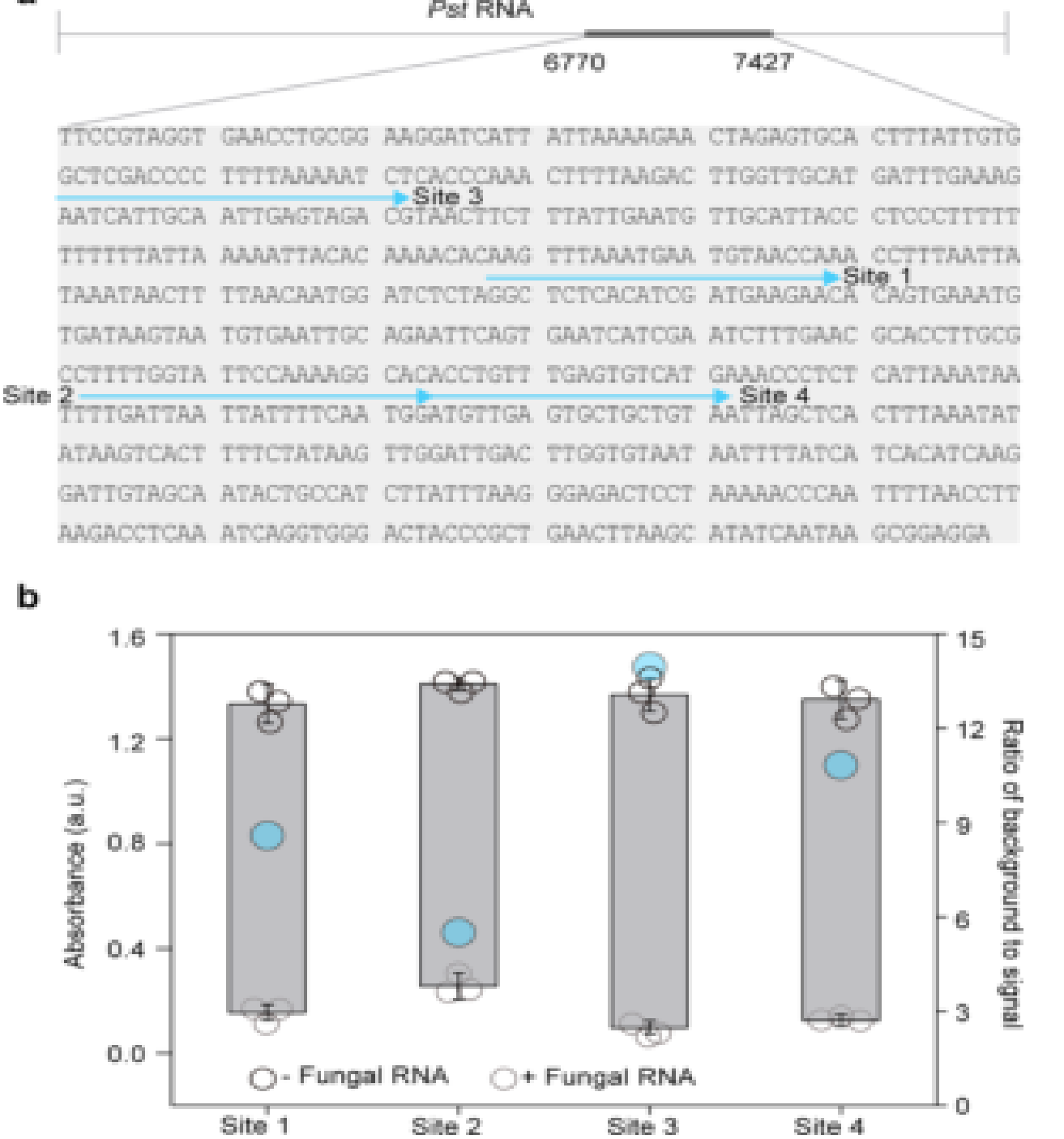
补充图3|Pst.ITS上RNA片段的可视化检测。
a.选择Pst ITS上4个长度为22nt的RNA片段作为靶位点。b.通过选择4个片段作为靶位点进行测定的吸光度响应。吸光度为在560nm的波长下测量。RNA片段、DProbe、尿素酶、尿素和酚红的浓度分别为200 nM、100 nM、1 nM、500 mM和250μM。b中的数据为平均值±标准差(n=3)。基于背景与信号吸光度的比率,使用该测定法,Pst的ITS中的结合位点3(6831-6852)产生最高的颜色变化,因此被选为靶RNA片段。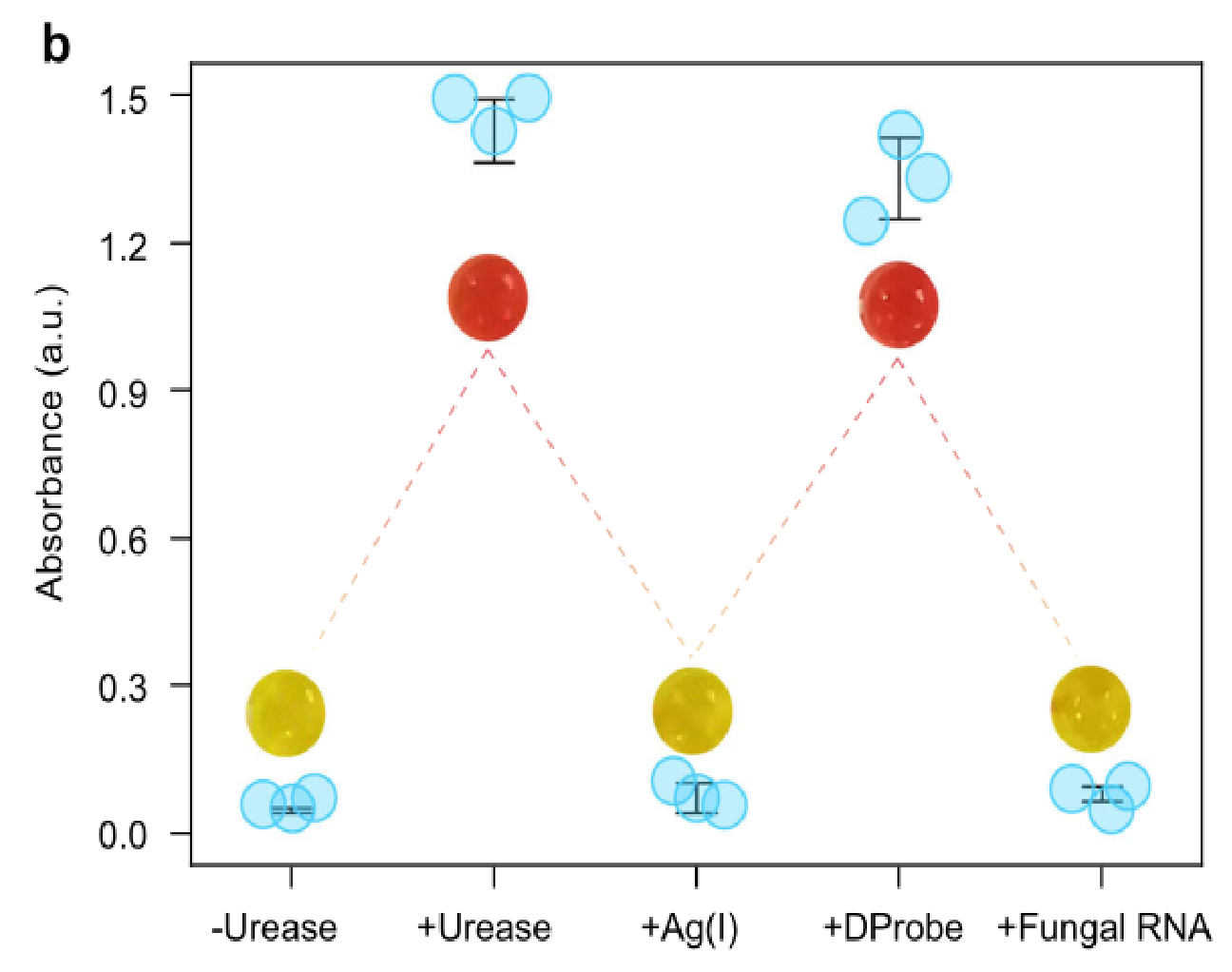
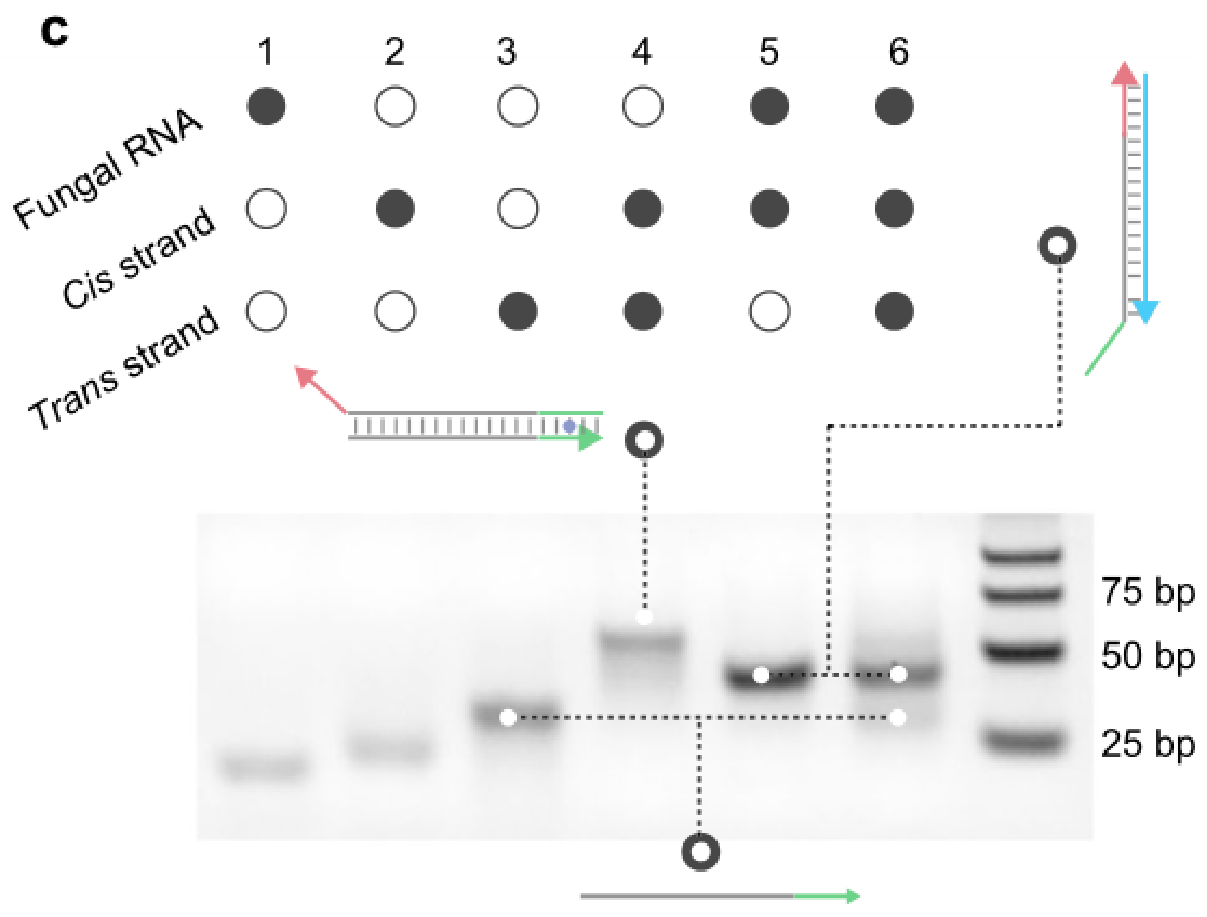
图4|b测定的每个反应在560nm处的吸光度 c每个反应的视觉结果
脲酶催化尿素水解产生氨,变红b DProbe、脲酶、尿素和酚红的浓度分别为100 nM、1 nM、500 mM和250μM。c TMSD反应的电泳分析。DProbe和真菌RNA的浓度分别为400nM和600nM。为了将DProbe与真菌RNA和电泳梯中顺式链的杂交体区分开来,在反式链的5′端添加了30nt poly-T。
补充图4|一种使用标记数据探针的TMSD方案
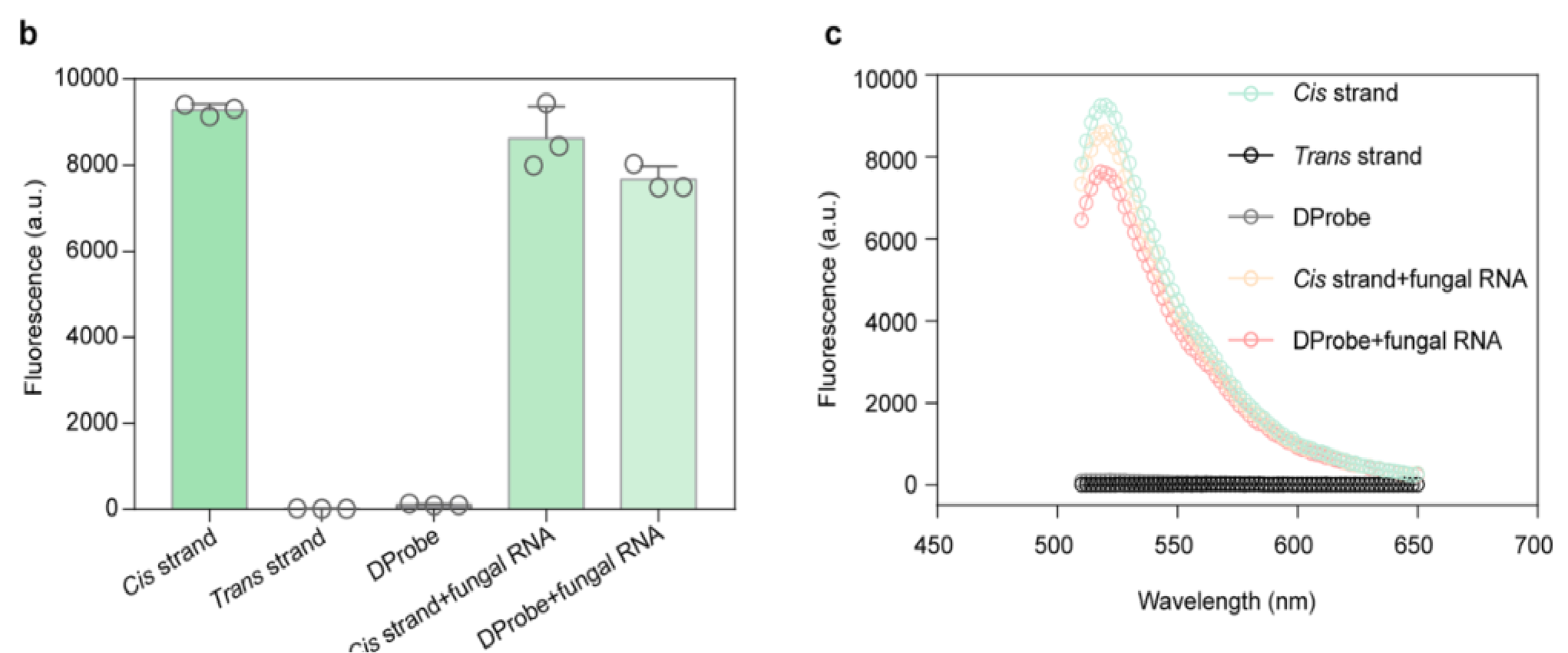
a 荧光团6-羧基荧光素(FAM)和猝灭剂黑洞猝灭剂-1(bhq 1)分别在顺式链的3’端和反式链的5’端进行修饰。通过顺式链和反式链的杂交形成d探针,导致FAM和 BHQ1的接近以及FAM的淬灭。真菌RNA与d探针前足趾的结合启动了 TMSD反应,并释放了d探针中的反式链,恢复了顺式链中标记的 FAM的荧光。b-C 参与TMSD反应的每个杂交体在520纳米的荧光强度(b)和在480纳米激发的光谱(c)。顺式链、反式链和真菌RNA的浓度为400纳米、400纳米和600纳米。b中的数据是平均值土标准差(n=3) 。单链顺式链(400nM)产生9281的荧光,并随着反式链的结合而淬灭,这表明双链 d探针的形成。真菌RNA序列的存在使d探针的荧光接近真菌RNA-顺式链杂交体的荧光,表明真菌RNA诱导了有效的链置换反应。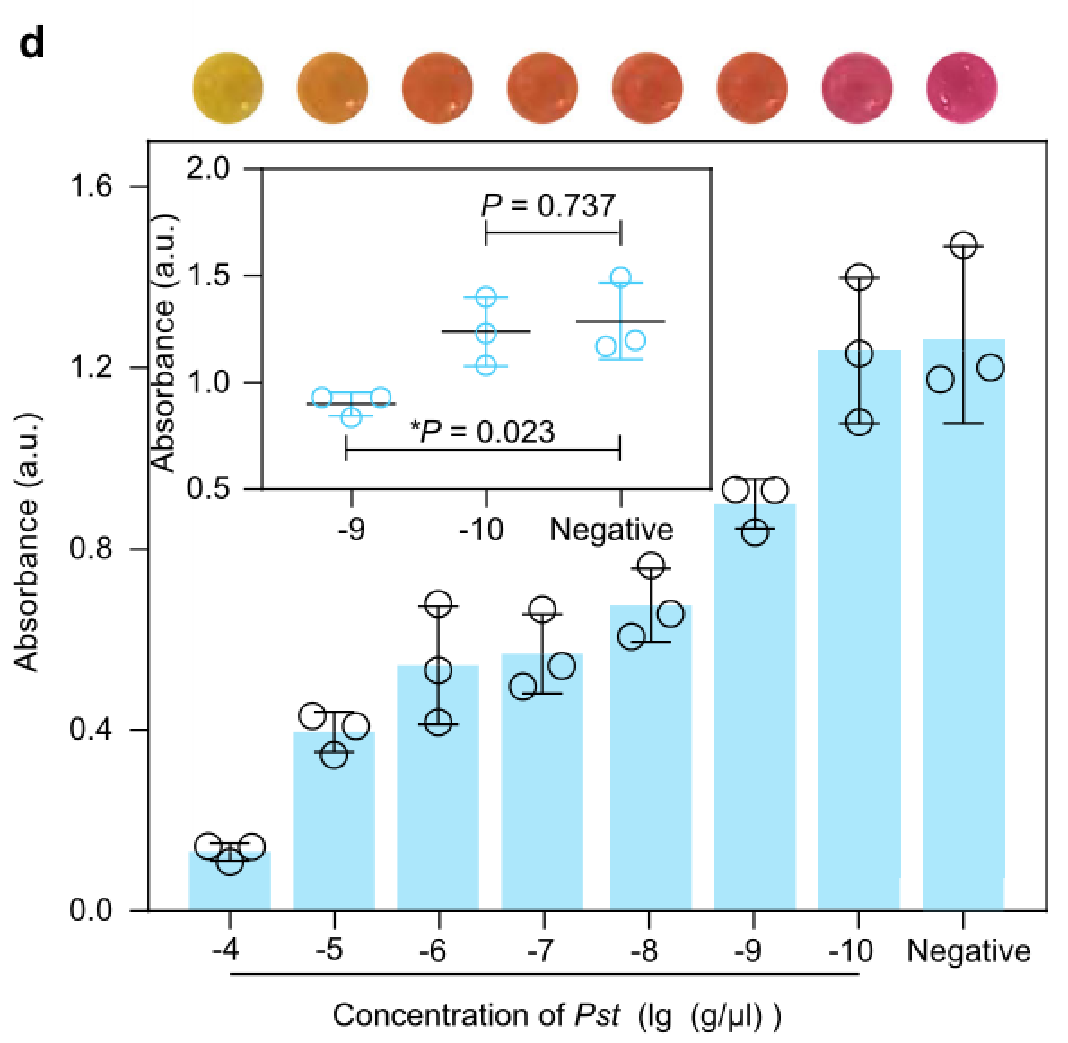
图5|每个样品的视觉结果(上图)和吸光度(下图)
lg(g/μl)表示Pst浓度的负对数。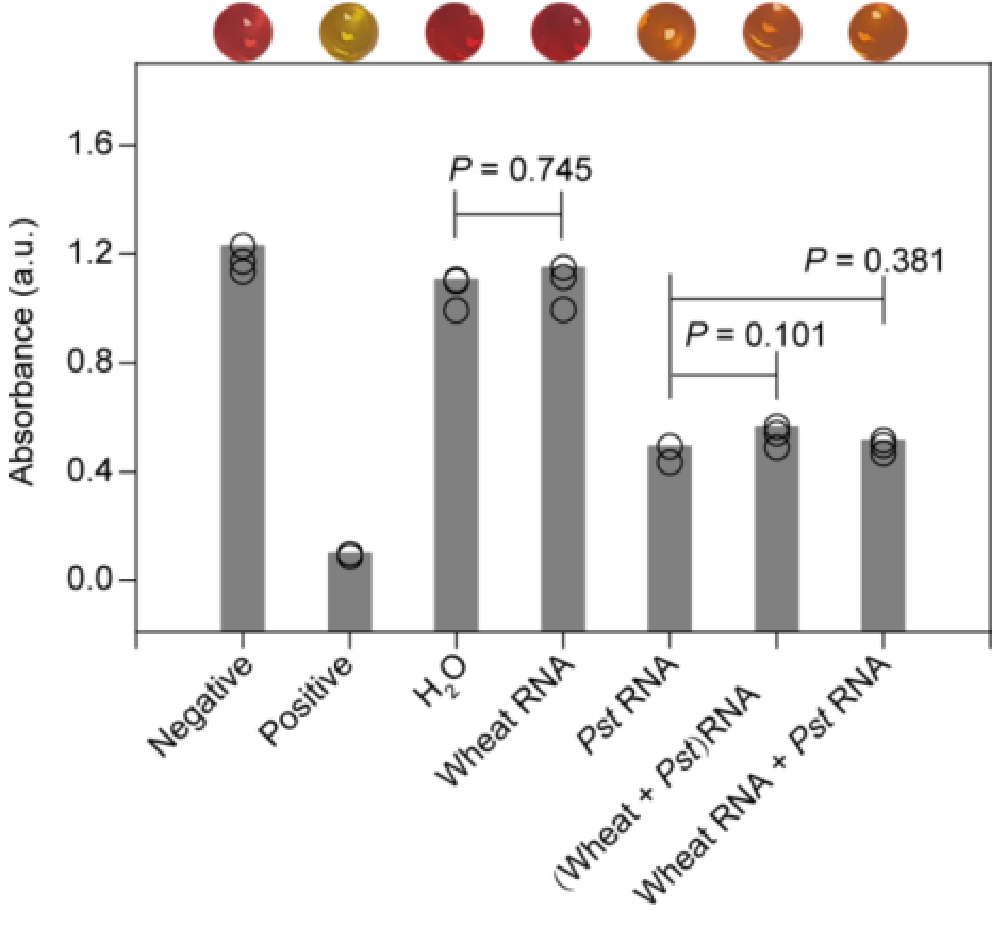
补充图5|小麦叶片RNA对Pst
在提取Pst感染的小麦叶片(标记为“(小麦+Pst)RNA”及其对照中,添加小麦RNA(标记为”小麦RNA+Pst RNA“)检测Pst的吸光度(底部)和视觉结果(上部)。对于样品“小麦RNA+Pst-RNA”,将10mg Pst孢子接种到面积为2×2cm(约20mg)的切割小麦叶片上使用Trizol试剂将载有Pst孢子的小麦叶片用于RNA提取。对于样品“小麦RNA+Pst RNA”,将从20mg小麦叶片提取的RNA加入到从10mg Pst孢子提取的RNA中。DProbe、脲酶、尿素和酚红的浓度分别为100 nM、1 nM、500 mM和250μM。2.2用比色纸多重检测真菌病原菌
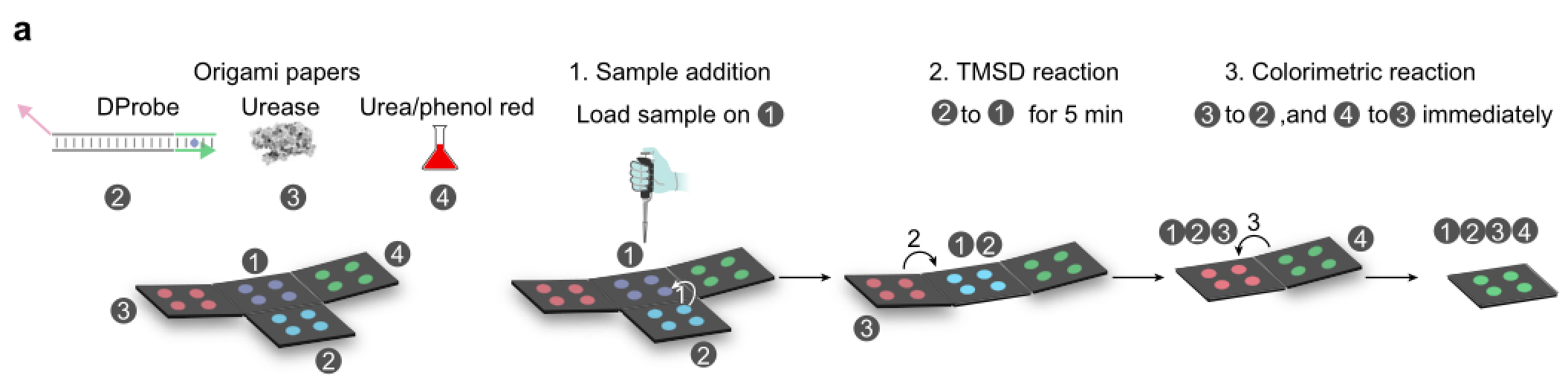
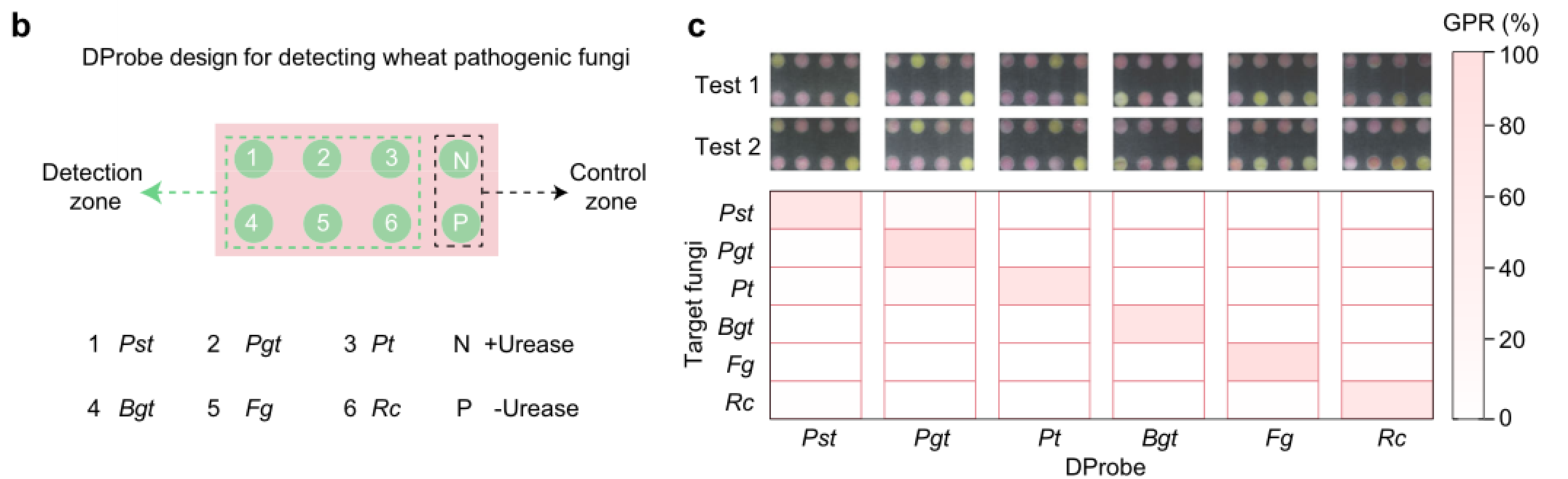
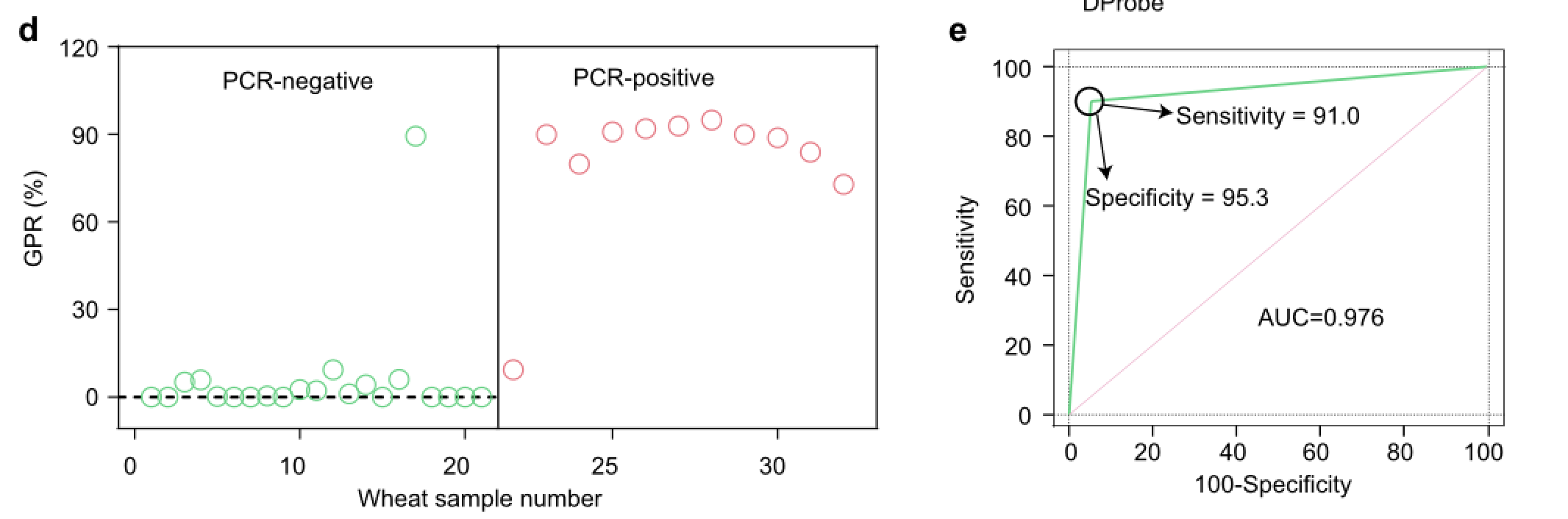
图6
a 使用纸模策略整合分析的每个反应。b 论文设计用于检测六种小麦病原真菌。c 使用比色纸对六种真菌进行多重检测。d 通过测试32个田间采集的小麦叶片样本获得的GPR值。e 检测结果比较Pst(磅/平方英尺)年32份小麦样品的感染情况(d日)使用比色测定法和qPCR。DProbe、脲酶、尿素和酚红的浓度为100 纳米,1 纳姆,500 mM和250 μM。通过设计八个样本点的折纸纸,六种致病真菌(Pst、Pgt、Pt、Bgt、Fg和卢比)可以并行检测。结果表明,使用一张装有同源探针的折纸,可以在无交叉干扰的情况下区分所有六种真菌与溶液中进行的比色分析相比,使用折纸进行比色分析产生了相当的灵敏度,并且允许检测低至1 纳克/微升Pst(磅/平方英尺)。
2.3真菌感染的早期诊断
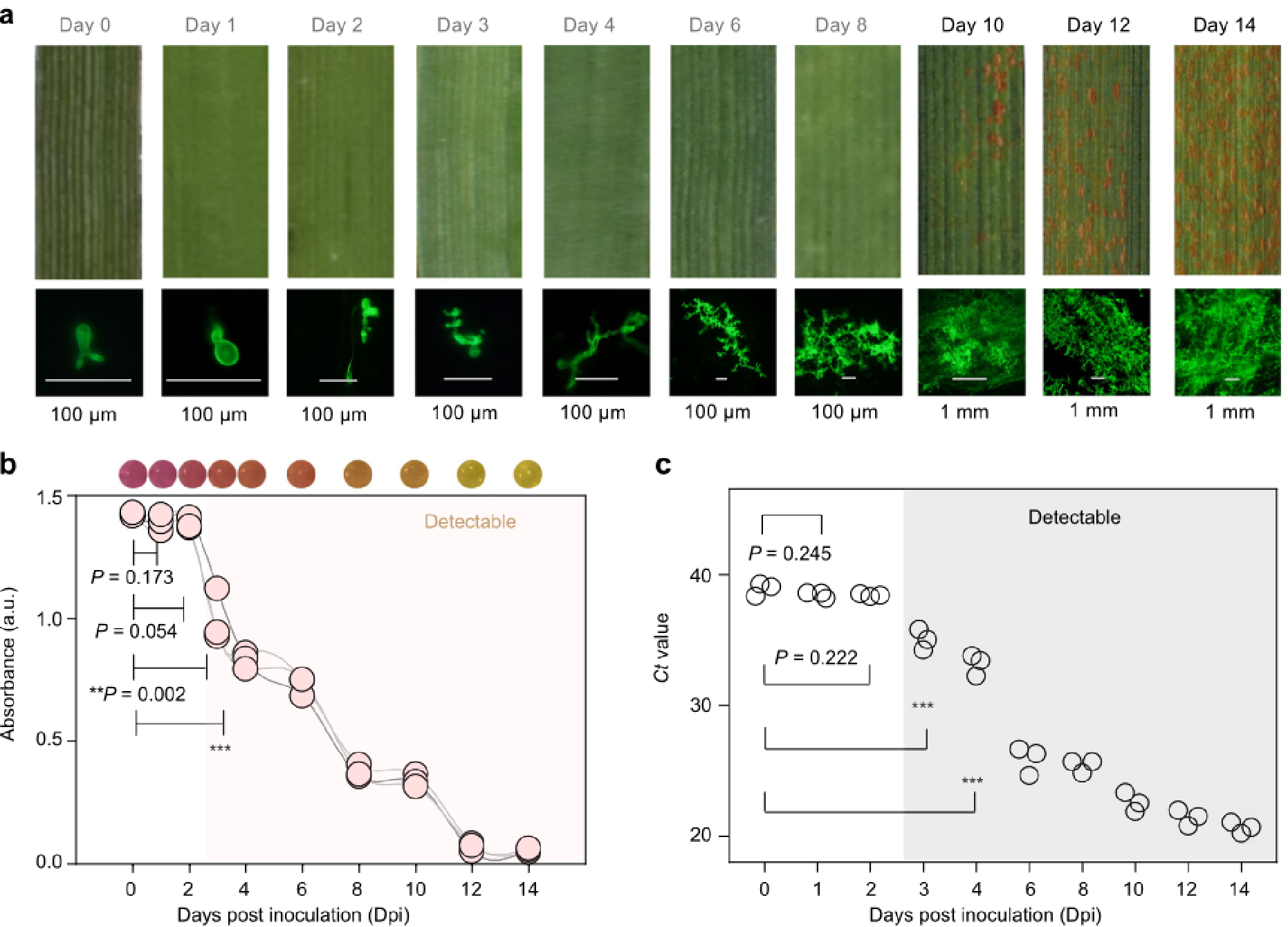
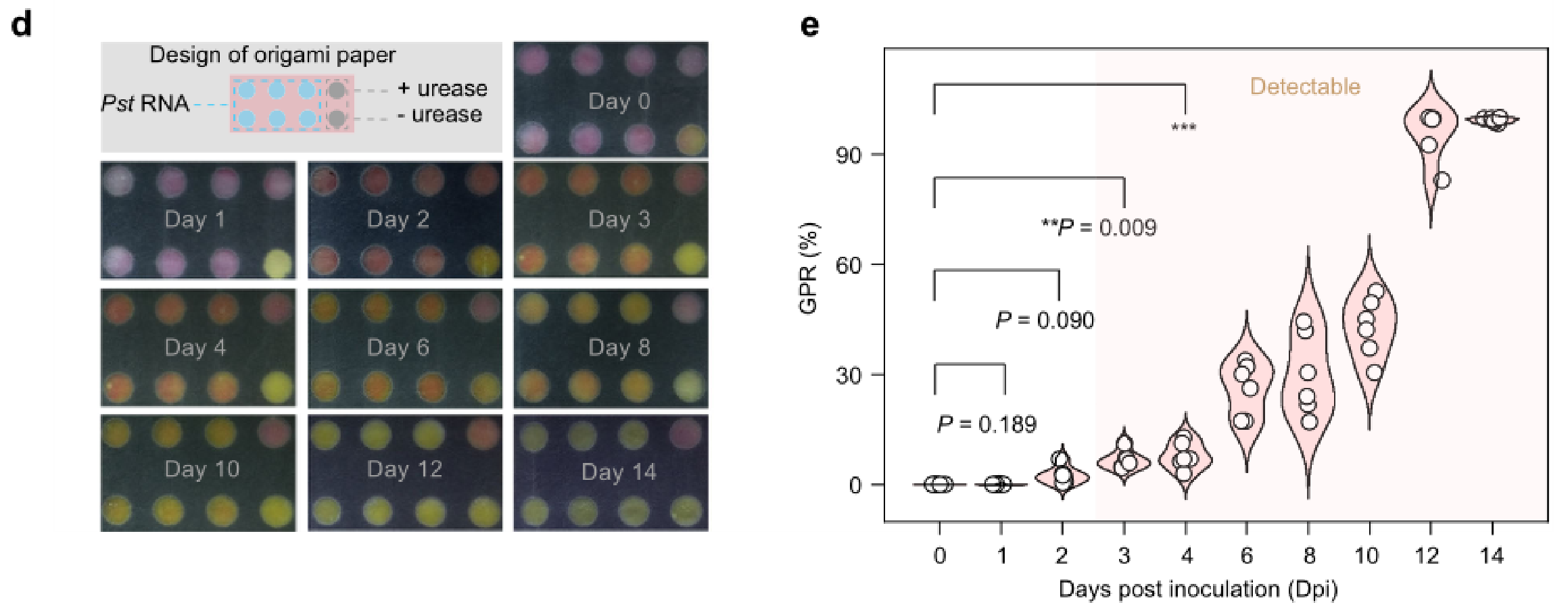
图7|真菌感染的早期诊断
a 小麦感染后14天叶片的表型分析Pst(磅/平方英尺)分离小麦叶片并使用智能手机拍照(上图),获得真菌结构的荧光显微照片(下图)。真菌结构用小麦胚芽凝集素染色。b 视觉结果(上部)和560时的吸光度 小麦叶片感染0~14天的检测结果为nm以下。c 检测Pst(磅/平方英尺)用qPCR检测感染0~14天的小麦叶片。d 并行检测的纸张设计Pst(磅/平方英尺)在受感染的小麦样本中。e 使用比色纸检测Pst(磅/平方英尺)小麦叶片感染1-14天。(小提琴图可以更好的反应数据的分布及概率密度,结合了箱式图及核密度图的优点。其内部是箱式图,外部是核密度图,某区域图形面积越大,某个值附近分布的概率越大。)总的来说,比色分析显示了早期检测Pst(磅/平方英尺)感染与qPCR相当,与症状观察相比,感染鉴定提前了7天。
2.4活菌检测有助于预测疾病的发生和严重程度
我们使用Pst(磅/平方英尺)含有活孢子和死孢子的混合物,其中活孢子的比例为0%、0.1%、1%、10%和100%(图8a ) 从等量的这些中提取核酸Pst(磅/平方英尺)混合物,并使用比色分析和qPCR进行分析。结果表明,在没有和存在小麦RNA基质的情况下,随着活菌数量的增加,吸光度信号逐渐减弱 。(图8b )
图8|活真菌的检测提高了疾病风险预测的精度
a 接种等量小麦的方案Pst(磅/平方英尺)含有0%、0.1%、1%、10%和100%活菌的混合物,以及感染14天后感染小麦叶片的症状观察。b-c 使用比色分析(b)和qPCR(c)检测a样品中的pst。 通过实现单核苷酸分辨率的田间核酸检测,推进了作物病害的分子诊断。应用涵盖病原真菌、病毒和细菌检测的田间诊断工具,有可能促进有效的作物病害管理,减少农药的使用,并通过在精确的植物诊断中减轻作物病害来促进可持续农业。
.参考文献:Zhang, T., Zeng, Q., Ji, F.et al.Precise in-field molecular diagnostics of crop diseases by smartphone-based mutation-resolved pathogenic RNA analysis.Nature communications,2023,14:4327.

